일반적으로 효소의 기능을 파악하는 과정 중에 이들이 촉진하는 생화학반응을 이해하는 것은 대사(metabolism) 시스템의 유전형-표현형 상관관계(genotype-phenotype association)를 파악하는 데에 매우 중요하다.
이는 응용기술에서도 중요한 데, 가령 특정 인체 세포의 게놈정보로부터 각 효소들이 어떤 생화학반응을 매개하는 지 정확하고 신속하게 알 수 있으면, 질병과 관련된 비정상적인 생화학반응들을 쉽게 발견하는 것이 가능해진다. 같은 맥락에서 여러 바이오제품을 생산하는 산업 생명공학에서도 효소가 관여하는 생화학반응을 정확히 알아낼 수 있는 기술은 제품 생산성을 높이는 데에 매우 중요하다.
특정 효소가 관여하는 생화학반응을 알 수 있는 가장 대표적인 방법은 해당 효소의 EC 번호(enzyme commission number)를 파악하는 것이다. EC 번호는 ‘EC 3.4.11.4’와 같이 효소가 매개하는 생화학반응들의 종류에 따라 총 4개의 숫자로 구성되어 있으며, 이 숫자들은 그 사이에 점으로 구분돼 있다.
중요한 점은 특정 효소에게 주어진 EC 번호를 통해서 해당 효소가 어떠한 종류의 생화학반응을 매개하는지 알 수 있다는 것이다. EC 3.4.11.4에서 ‘EC 3’은 가수 분해 효소를, ‘EC 3.4’는 펩타이드 결합에 작용하는 가수 분해 효소를, ‘EC 3.4.11’은 폴리펩타이드의 말단 아미노산을 자르는 가수 분해 효소를, ‘EC 3.4.11.4’는 트라이펩타이드의 말단 아미노산을 자르는 가수 분해 효소를 뜻한다.
지난해까지 그동안 EC 번호를 예측해주는 컴퓨터 방법론들이 최소 10개 이상 개발됐다. 그러나 이들 모두 예측 속도, 예측 정확성 및 예측 가능한 EC 번호 범위 측면에서 더욱 발전의 여지가 있었다. 특히 현대 생명과학 및 생명공학에서 이루어지는 연구의 속도와 규모를 고려했을 때, 이들 방법론의 성능은 충분하지 않았다.

이 가운데 국내연구진 KAIST(총장 신성철) 생명화학공학과 이상엽 특훈교수와 김현욱 교수의 초세대 협업연구실 공동연구팀이 인공지능 딥러닝(deep learning) 기술을 이용해 효소의 기능을 신속하고 정확하게 예측할 수 있는 컴퓨터 방법론 DeepEC를 개발한 것이다.
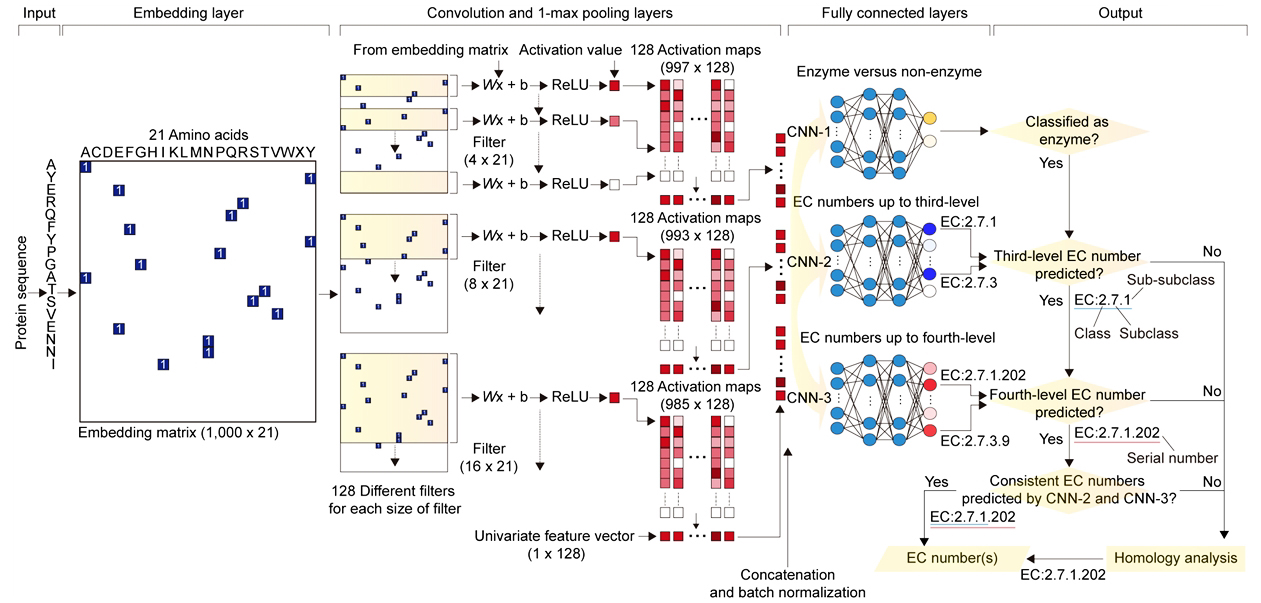
이번 연구팀이 개발한 DeepEC는 효소 단백질 서열의 EC 번호를 빠르고 정확하게 예측해 주는 컴퓨터 방법론으로서, 1,388,606개의 단백질 서열과 이들에게 신뢰성 있게 부여된 EC 번호가 담긴 바이오 빅데이터에 딥러닝 기술을 적용하여 개발한 것으로 DeepEC는 주어진 단백질 서열의 EC 번호를 예측하기 위해서, 3개의 합성곱 신경망(Convolutional neural network)을 주요 예측 기술로 사용하며, 합성곱 신경망을 통해서 EC 번호를 예측하지 못했을 때는 서열정렬(sequence alignment)을 통해서 EC 번호를 예측하게 된다.
특히, DeepEC에서 첫 번째 합성곱 신경망은 주어진 단백질 서열이 효소에 해당하는지를 판단하며, 두 번째 합성곱 신경망은 세 번째까지의 EC 번호 숫자를, 마지막 세 번째 합성곱 신경망은 네 개의 온전한 EC 번호 숫자들을 예측한다. DeepEC가 EC 번호 예측을 이들 세 개의 합성곱 신경망을 통해서 진행할 경우, 이들 합성곱 신경망이 모두 일관된 결과를 예측할 때에만 최종 결과를 만들어내는 것이다.
또한 DeepEC의 성능을 평가하기 위해서, 이전에 발표된 5개의 대표적인 EC 번호 예측 방법론과 비교한 결과, DeepEC가 가장 빠르고 정확하게 주어진 단백질의 EC 번호를 예측하는 것으로 나타났다. 이 비교 분석에서는 총 6개의 EC 번호 예측 방법론 개발 시 전혀 사용된 적이 없는 201개의 효소 단백질 서열, 6개의 EC 번호 예측 방법론 개발 시 모두 사용된 적이 있는 2,310개의 효소 단백질 서열, 본 연구의 학습데이터로부터 무작위로 선정된 1,600개의 효소 단백질 서열 및 현재까지 완료된 9,513개의 게놈서열로부터 얻을 수 있는 총 33,942,253개의 단백질 서열 등이 활용되었다.
이 밖에 DeepEC가 5개의 다른 EC 번호 예측 방법론 대비 단백질 서열의 도메인(domain)과 기질 결합부위 잔기(binding site residue)에 변이를 인위적으로 주었을 때, 가장 민감하게 그 영향을 감지할 수 있는 것으로 나타났다. 또한 DeepEC를 인체 대사 컴퓨터 모델링에 적용해 본 결과, 이미 실험증거가 있음에도 이전 인체 대사모델에는 제대로 반영이 안 된 212개의 생화학반응을 새로이 발견한 것으로 KAIST 초세대 협업 연구실 통해 이뤄낸 이번 성과는 대표적인 모범사례 될 것으로 기대를 모으고 있다.
김현욱 교수는 “DeepEC의 성능을 평가하기 위해서 이전에 발표된 5개의 대표적인 EC 번호 예측 방법론과 비교해보니 DeepEC가 가장 빠르고 정확하게 주어진 단백질의 EC 번호를 예측하는 것으로 나타났다”라며 “효소 기능 연구에 크게 이바지할 것으로 기대한다”라고 말했다.
또한 이상엽 특훈교수는 “이번에 개발한 DeepEC를 통해서 지속해서 재생되는 게놈 및 메타 게놈에 존재하는 방대한 효소 단백질 서열의 기능을 보다 효율적이고 정확하게 알아내는 것이 가능해졌다”라고 말했다.
한편, 공동연구팀의 류재용 박사가 1 저자로 참여한 이번 연구결과는 국제학술지 ‘미국 국립과학원 회보(PNAS)’ 지난 6월 20일 자 온라인판(논문명: Deep learning enables high-quality and high-throughput prediction of enzyme commission numbers/ 논문 링크)에 게재됐다.


